eDESIGNERを使ってみる(前処理編)

前回の記事から大分間が空いてしまいましたが、引き続きオープンソースツールのeDESIGNERの使い方を書いていきます。今回は、eDESIGNERで使う試薬(Building Block, BB)ファイルはどのような形で用意すれば良いかという所までを扱います。eDESIGNERの概要と環境構築については前記事をご覧ください。
tky-pychemweb.hatenablog.com
なお、前回の環境構築はLinuxで行いましたが、eDESIGNER自体はpythonのみで動作し、M1チップのMBAでも動作確認が取れたため、スクショはMacで撮っています。
eDESIGNER全体の流れ
eDESIGNERが行う処理の全体の流れを下図に示します。

ライブラリーを構成するBBのリストからスタートし、含まれる化合物の全体構造を得ることをゴールとすると、eDESIGNERが担当するのは、アノテーション付きのBBファイルを入力にして、ライブラリーを構築するレシピのような設定ファイルを出力するところまでになります。その前後に当たるアノテーション付きのBBファイルの準備と構造発生で、LillyMolが必要になるところが使うのを難しくしている要因です。そしてお試しのサンプルファイルもないんですよね。。この「アノテーション付きのBBファイル」というのは、どのようなファイルをどうやって準備すれば良いのか、というのが今回のメインテーマです。
アノテーション付きのBBファイル
アノテーションとは何ぞやという点ですが、各BBがどのような官能基をいくつ持っているかを数値の羅列で表現したファイルになります。何の官能基を持っているかを調べてくださいというのが事前に定められており、それに従ってファイルを準備する必要があります。さらに、eDESIGNER処理で利用するID、分子量、回転可能結合数も追記しています。このファイルを準備することが、今回のゴールになります。
アノテーション付きファイルの作成
入力BBファイル
まず、入力となるBBのファイルをSMILES形式で準備します。この段階では分類などをする必要はなく、ただ使うかもしれないBBを全て含むファイルを作れば大丈夫です。RDKitで読み込めないと今後エラーが出るので、この段階でRDKitで準備しておくとスムーズでしょう。このファイルをsample_bb.smiというファイルとします。
tsubstructure
元論文のSupplementary informationをよく読むとLillyMolのtsubstructureというツールを使うと書かれています。>
このまま使うとうまくいかない部分があるため、オプションを追加して以下コマンドを実行します。username部分は各自のインストールフォルダに合わせて変更してください。
tsubstructure -v -u -q F:/home/<username>/github/LillyMol/contrib/data/DEL/AnnotationQueries/queries_del -A D -a ./sample_bb.smi > ./sample_annot.txt
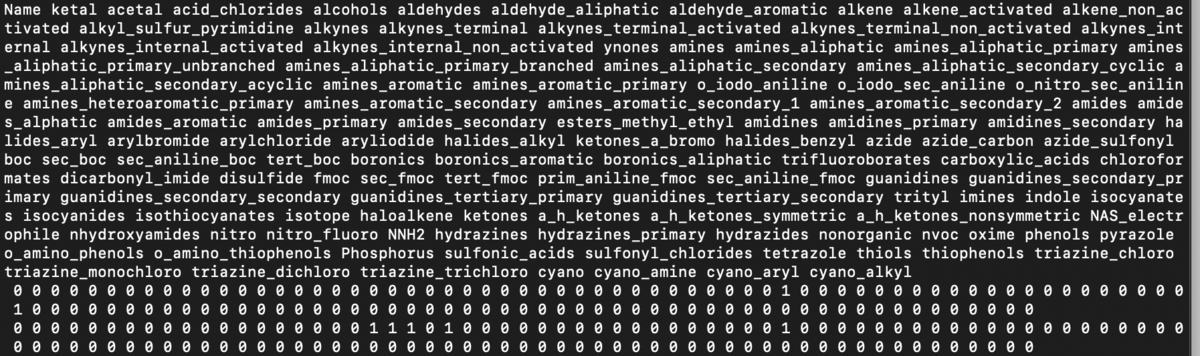
この結果出力されるsample_annot.txtは以下のようなファイルになります。40-50の定義された官能基を各BBがいくつ有しているかを羅列したファイルが出来上がります。
統合ファイルの作成
最後に、IDといくつかの物性値を追加して、eDESIGNERのインプットに使うファイルを作成します。sample_bb.smiとsample_annot.txtを統合するpythonスクリプト(prepare_bbt_input.py)を作成して処理します。
python prepare_bbt_input.py sample_bb.smi sample_annot.txt
スクリプトの中身は以下のようになっています。
import sys import pandas as pd from rdkit import Chem from rdkit.Chem import AllChem, Descriptors def calc_props(smiles_list): id_list = [] mw_list = [] rotb_list = [] for idx, smi in enumerate(smiles_list): bbid = f"test_{idx}" mol = Chem.MolFromSmiles(smi) mw = Descriptors.MolWt(mol) rotb = Descriptors.NumRotatableBonds(mol) id_list.append(bbid) rotb_list.append(rotb) mw_list.append(mw) prop_df = pd.DataFrame({"ID": id_list, "SMILES": smiles_list, "ROTB": rotb_list, "MW": mw_list}) return prop_df def merge_annot(prop_df, annot_file): df_annot = pd.read_csv(annot_file, sep=" ") df_annot = df_annot.drop(columns=["Name"]) if df_annot.shape[0] == prop_df.shape[0]: df = pd.concat([prop_df, df_annot], axis=1) else: print("Number of rows is not the same.") exit() df.to_csv("BBT_input.txt", index=False, sep=" ") if __name__ == '__main__': args = sys.argv if len(args) == 3: smiles_file, annot_file = args[1], args[2] smiles_list = [] with open(smiles_file, 'r') as inp: for smi in inp.readlines(): smiles_list.append(smi.rstrip()) prop_df = calc_props(smiles_list) merge_annot(prop_df, annot_file) else: print("Usage: python prepare_bbt_input.py smiles_filename annotation_filename") exit()
calc_propsでIDと物性値を生成し、 merge_annotでアノテーション情報を統合してBBT_input.txtというファイルに出力します。
完成した入力ファイル
以上を実行した結果得られるBBT_input.txtは以下のようなファイルになります。なお、ID部分のカラム名はIDにしておかないと後々エラーが出るようです。次の記事では、いよいよこのファイルを使ってeDESIGNERを動かしていきます。
