NIHのDELウェビナーを見た

以前DELの総説的なウェビナーがあるということを教えてもらったのですが、遅ればせながら見てみました。ウェビナー自体は2021年にNIH主催で開催されたもので、動画のアーカイブが公開されています。二年ほど前の内容になりますが、分野の第一人者や経験豊富なメガファーマ、スタートアップの面々が演者に名を連ねており大変勉強になりました。セッションは3部制で、Chemistry, Selection, Data Analysisから成ります。私は分野的にData Analysisのパートを視聴し、参考になった点を備忘録的にメモします。フルの動画は以下サイトに公開されていますのでご参考ください。
NCATS — AGM Workshop on DNA-Encoded Libraries for Lead Discovery
- Beyond the Cube: Informatics Methods for Best Practices in DEL Analysis (Day2 1:00-1:40)
- DEL Screen Triage Strategies Toward Tractable Chemical Matter (Day2 1:41-2:28)
- Strategies for Hit to Lead Optimization: Common Tactics Applied to DNA-Encoded Library (DEL) Derived Hits (Day2 2:30-3:06)
- DNA Encoded Libraries and Interpretable ML for Hit Triage (Day2 3:44-4:07)
Beyond the Cube: Informatics Methods for Best Practices in DEL Analysis (Day2 1:00-1:40)
Nurix社はアメリカのスタートアップで、PROTACを作る際の標的タンパク(POI)に結合する側の化合物を取得する際にDELを活用しています。かなり多くのセレクションをこなしている様子で、かなりシステム化されているように感じました。主にNGS後の解析、offDNA合成する化合物の選択についての発表でした。
DEL Screen Triage Strategies Toward Tractable Chemical Matter (Day2 1:41-2:28)
ファイザーのDELプラットフォーム責任者と思われる方の発表で、スクリーニングしてヒット候補をプロジェクトチームに引き渡すことをミッションとしているチームのようです。専門部隊がいることがメガファーマらしさを感じさせます。データをDB化してDEL_DVと名付けたSpotfireビューで可視化する流れまで構築されており、将来像として大変参考になりました。ここまでするのは専門じゃないと苦しいなあというのはありますが、、
- ライブラリーは数を犠牲にしても物性を良くする方向に向かっている
- MPOスコアはカットオフではなくカラーリング用途で使っている
- 一つの標的タンパクに対して10前後のセレクションを実施して複合的に解析するのが標準的
- ビーズのみ、ポジコンあり、複合体タンパクありなどのセレクションを加えるのが代表的
- セレクション条件が多くなると解釈が困難になるため、プロファイルでクラスタリングする
- 化合物レベルまたはline featureレベルでリード数/ブランクでのリード数を算出して解析する
- Spotfireは元データが大量なためオンデマンドロードする仕様
- ヒットからの合成展開については以下文献が参考になると紹介されていた
- BALI-MSによるonDNAでのバインダー判定をoffDNA合成前に実施する
- onDNAでバインダー確認できるのは43%程度で、その内74%が目的物がバインダー、26%はバイプロがバインダー
Strategies for Hit to Lead Optimization: Common Tactics Applied to DNA-Encoded Library (DEL) Derived Hits (Day2 2:30-3:06)
GSKのDELチームのGang Yao氏から、主にoffDNA後にフォーカスし、報告されているDEL由来のヒットからリードへの軌跡を紹介する内容です。ファイザーのBALI-MS報告後に、GSKはcleavable linkerを使ったon-DNAによるヒット同定法を提唱しているそう。
- 意外とDNAタグとBB1をtruncateしたものが初期ヒットになっている
- 成功した最適化は全てLLEを改善する方向に向かっており、基本脂溶性を下げる
- SARがブロードなBBは削っても良いという考えのようで、活性下がってもLLE上がれば問題なし
- BRD4の例ではフラグメントヒットパーツとのマージも利用
MW 400-500からスタートしている例も結構あり、実質2cycleくらいのサイズでも良いのでは感は感じました。
DNA Encoded Libraries and Interpretable ML for Hit Triage (Day2 3:44-4:07)
GSKから、毒性評価に使われているPDE3AのQSARモデルのApplicability Domainを改善できるケモタイプをDELから探す試み。DELのセレクション結果から二分類モデルを作り、Chemblのデータセットを予測したが精度は今一つな様子だった。
文献記載化合物の構造式を取得する
論文や特許に記載されている、パブリックなデータを使って何らかの検証やソフトウェアのデモなどを行いことがあるかと思います。私自身も先日そのような状況に遭遇し、とある文献をピックアップしました。SAR表に書かれている構造式を全て書き起こすのは流石に大変なので、どうやって抜き出すのが良いか調べた小ネタメモです。結論から言うとChEMBL様様という話です。
文献から探す場合
まず、含まれる構造式を抜き出したい、または構造情報の有無を調べたい文献が定まっている場合です。
- ChEMBLトップページで論文のDOIを検索
- Documentに付与されたChEMBL IDをクリック
- Document Report Card下方のCompound SummariesにあるAssociated Compounds for Document ... をクリック
- 化合物一覧が表示されるので必要なものを選択してCSV or TSVボタン押下
- ツールバーすぐ下の分かりにくい場所に出るhereを押してダウンロード
2023/07時点では活性値は出力されないようで、別途化合物のChEMBL IDから取得する必要がありそうです(私のケースではIDとSMILESの構造で事足りました)。CSVと言いつつコンマ区切りではなかったので要注意です。また、複数選択時のSDF ボタンはバグがあるようで、構造が分かれて出力されませんでした。ひとまず、テキスト系で書き出して、SMILESから変換することでSDFを得られました。
化合物名から周辺誘導体情報を探す場合
臨床まで進んだ化合物などで、興味のある化合物に化合物名がついており、その探索段階で作られた類似構造の化合物に辿り着きたいような場合です。
- ChEMBLトップページで化合物名を検索
- 構造と化合物名が表示されるので望みのものであれば選択
- Compound Report Card下方のLiteratureでDocuments by Yearを選択
- 合成展開されていそうな文献のChEMBL IDを選択して、文献から探す場合の1へ
周辺化合物を網羅的に調べられるわけではありませんが、ChEMBLにあって簡便に抽出できそうかどうかは判断できると思います。
おわりに
正直他にいい方法が色々ありそうな気がしますので、情報お待ちしています。
DELに含まれる化合物の全体構造を発生させる

前回までの記事では、eDESIGNERというオープンソースのツールを使って、DELのデザインを行ってきました。しかし、実際にデザインしたDELにどのような構造の化合物が含まれているかは確認できていませんでした。一つのDELでも数千万〜数億の構造を含むことも珍しくないため、全構造を発生させる必要性は低いですが、いくつかサンプリングして構造発生できると、物性値の分布なども確認できて便利です。eDESIGNERで設計したライブラリーの設定ファイルを基に、構造発生する方法をまとめます。
前提条件
以下の環境が整っているとして進めます。詳細は以前の記事の該当部分をご確認いただければと思います。
- 以下レポジトリをクローンし、eDESIGNERを実行してlibrary configファイルが作成できている
- LillyMolのインストールが完了し、各種機能がコマンドで実行できる(パスが通っている状態)
- eDESIGNER及びLillyMolが使用できるconda環境を立ち上げている
githubからcloneしたedesigner_core/edesigner/下で作業し、前提条件を満たしていれば、resultsフォルダにconfigファイルが出力されているはずです。
% tree results results ├── 20230428_3Cboth34_config.txt ├── 20230428_3Cboth34_libDESIGNS.pic
サイクル毎BBファイルの作成
全体構造を発生させるためのレシピに該当するのはconfigファイルになりますが、材料となるBBのファイルはe_bb_retriever.pyを使って作成することができます。サイクル毎に材料となるBBがcsvで出力されます。
# 出力先のフォルダを準備 % mkdir design1 % python e_bb_retriever.py -tk 20230428_3Cboth34 -df 1 -of design1 e_bb_retriever.v.9.0.0 Loading pickled designs to object... Creating dataframes and dumping them into files... OK % tree -F design1 design1/ ├── 20230428_3Cboth34_1_C1_all.csv ├── 20230428_3Cboth34_1_C1_int.csv ├── 20230428_3Cboth34_1_C2_all.csv ├── 20230428_3Cboth34_1_C2_int.csv ├── 20230428_3Cboth34_1_C3_all.csv ├── 20230428_3Cboth34_1_C3_int.csv
各オプションの意味は以下の通りです。
- df : design number 今回は1を指定
- of : 出力先フォルダ名
所望のライブラリー設計ファイルの切り出し
これは提供されているスクリプトではできないので、ユーティリティスクリプトを準備しました。edesigner/ext_utils/フォルダ下にありますので、そこに移動して以下のように実行します。引数は全て必須となっており、何番の設定を抜き出すか事前に調べる必要があります。また、-ofはpar.parのfinal_compounds_folderに記載した場所と一致させる必要があります。
% cd ext_utils % python extract_config.py -tk 20230428_3Cboth34 -df 1 -bf design1 -of bbt Number of libDESIGNs 72 % tree ../results ../results ├── 20230428_3Cboth34_1_int_config.txt << 作成された ├── 20230428_3Cboth34_config.txt ├── 20230428_3Cboth34_libDESIGNS.pic └── sample_generator.log % tree ../bbt ../bbt ├── C1.int.smi ├── C2.int.smi └── C3.int.smi
resultsに抜き出した設定ファイル、-ofに指定したbbtに各サイクルで使われるBBファイルが出力されます。
次に使うmolecular_transformsは、なぜか上記で出力されたBBのCSVに対応していないため、修正してC1.int.smi等のファイルに出力し直しています。
全体構造の生成
材料が揃いましたので全体構造を発生させてみましょう。構造発生にはLillyMolのmolecular_transformsを使って以下のようにします。
# Move to /edesigner_core/edesigner/ % cd ../ # 結果出力用のフォルダを準備 % mkdir product % molecular_transformations -N 10000 -u -d -v -S ./product/prod -m RMX -z i -M RMX -Z -W rxsep='>>' -W rgsep='+' -A D -T results/20230428_3Cboth34_1_int_config.txt Aromaticity done by Daylight rules Will suppress duplicate molecules - within current generation Reagents not matching the query will be ignored Molecules not reacting will be ignored Will discard scaffolds that show multiple substructure search hits Will produce 10000 random selections Read 39 records from control file Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified Sidechain_Reaction_Site::construct_from_msi_object: no joins specified 3 reactions in group 0 1 reactions in group 1 4 reactions in group 2 1 reactions in group 3 11 reactions in group 4 2 reactions in group 5 2 reactions in group 6 2 reactions in group 7 2 reactions in group 8 2 reactions in group 9 2 reactions in group 10 Ran 100000 iterations to produce 192 random molecules. Skipped 91552 duplicates at top level, 91552 subsequent dups Read 0 molecules, changed 1 2816 hits to query 0 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_amines_AND_carboxylic_acids' 2816 hits to query 1 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_amines_AND_carboxylic_acids' 2816 hits to query 2 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_amines_AND_carboxylic_acids' 8448 hits to query 3 '6.1.1_N-Boc_deprotection_FROM_boc_AND_Null' 2112 hits to query 4 '9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic' 2112 hits to query 5 '9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic' 2112 hits to query 6 '9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic' 2112 hits to query 7 '9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic' 8448 hits to query 8 '9.7.61_Ester_hydrolysis_FROM_esters_methyl_ethyl_AND_Null' 768 hits to query 9 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 10 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 11 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 12 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 13 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 14 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 15 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 16 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 17 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 18 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 768 hits to query 19 '2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines' 1536 hits to query 20 '6.1.1_N-Boc_deprotection_FROM_boc_AND_Null' 4224 hits to query 21 'NOOP' 768 hits to query 22 '6.1.1_N-Boc_deprotection_FROM_boc_AND_Null' 4224 hits to query 23 'NOOP' 0 hits to query 24 '6.1.6_N-Fmoc_deprotection_FROM_fmoc_AND_Null' 4224 hits to query 25 'NOOP' 0 hits to query 26 '6.1.6_N-Fmoc_deprotection_FROM_fmoc_AND_Null' 4224 hits to query 27 'NOOP' 384 hits to query 28 '9.7.61_Ester_hydrolysis_FROM_esters_methyl_ethyl_AND_Null' 4224 hits to query 29 'NOOP' 192 hits to query 30 '9.7.61_Ester_hydrolysis_FROM_esters_methyl_ethyl_AND_Null' 4224 hits to query 31 'NOOP' Wrote 192 molecules % tree product/ product/ └── prod.smi % head -n 2 product/prod.smi [NH]=C(N[C@H](Cc1[n]cccc1)C(=O)N1C(C(=O)NC)CN(C(=O)OC(C)(C)C)CC1)N1C(C(=O)N(CCO[13CH3])C)(CCCC1)C core+10:1_Test:test_693>>2.1.2_Carboxylic_acid_+_amine_condensation_FROM_amines_AND_carboxylic_acids>>6.1.1_N-Boc_deprotection_FROM_boc_AND_Null+11:1_Test:test_532+C=N>>9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic>>9.7.61_Ester_hydrolysis_FROM_esters_methyl_ethyl_AND_Null+10:1_Test:test_694>>2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines OCC1CN(C)CCN(C(=O)[C@@H](NC(=[NH])N2CC(C(=O)N(CCO[13CH3])C)(CCC2)C)Cc2[n]ccc[n]2)C1 core+10:1_Test:test_682>>2.1.2_Carboxylic_acid_+_amine_condensation_FROM_amines_AND_carboxylic_acids>>6.1.1_N-Boc_deprotection_FROM_boc_AND_Null+11:1_Test:test_531+C=N>>9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic>>9.7.61_Ester_hydrolysis_FROM_esters_methyl_ethyl_AND_Null+10:1_Test:test_474>>2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines
オプションが沢山ありますが、-Sで出力先と接頭語、-Tで抜き出した設定ファイルを指定する事以外はそのままでいいと思います。これで、DELを構成する化合物から、ランダムに選ばれた10000化合物の全体構造のSMILESがファイルに出力されます。出力ファイルは全体構造SMILES space どのように作ったかが記載された、ゴチャゴチャしたファイルになります。
代表的な記述子の算出
論文中で述べられているような記述子を計算するスクリプトも用意されていない(と思う)ので、ext_utilsフォルダにスクリプトを用意しました。以下のように生成物のフォルダを引数として指定すると、中にある拡張子smiのファイルを読み込んで記述子を計算します。計算する項目はMW, tPSA, LogP, 芳香環数、回転可能結合数、重原子数、fsp3です。実行したフォルダにprod_descs.txtというファイルが出力されます。
% cd ext_utils/ % python desc_calculation_multi.py ../product/ 192 compounds will be processed. All the structures were successfully calculated calculation time 0:00:00.354224 % head -n 2 prod_descs.txt SMILES MW TPSA LogP AROM ROTB HAC FSP3 [NH]=C(N[C@H](Cc1[n]cccc1)C(=O)N1C(C(=O)NC)CN(C(=O)OC(C)(C)C)CC1)N1C(C(=O)N(CCO[13CH3])C)(CCCC1)C 631.783160.5 1.06 1 9 45 0.677
これで気になるDELについては、全体構造を発生させて、計算物性値の確認をすることができるようになりました。物性値を意識したDELを作っていくのも一案ではないでしょうか。
eDESIGNERを使ってみる(番外編)

前回まででeDESIGNERのメインの機能は使い終わりましたが、全スクリプトの半分ほどしか使っていませんでした。他のスクリプトはどんな機能があるのか見ていきましょう。
e_bb_retriever.py
主要スクリプト以外で、おそらく一番重要なスクリプトです。ライブラリーconfigファイルに出力された各ライブラリーの構造発生に使うBB構造を集めてくれるスクリプトです。使い方は以下の通りで、dfに構造発生したいライブラリーの番号を指定します。ofは結果が出力されるフォルダです。
% python e_bb_retriever.py -tk 20230428_3Cboth34 -df 1 -of design1 e_bb_retriever.v.9.0.0 Loading pickled designs to object... Creating dataframes and dumping them into files... OK % tree -F design1 design1/ ├── 20230428_3Cboth34_1_C1_all.csv ├── 20230428_3Cboth34_1_C1_int.csv ├── 20230428_3Cboth34_1_C2_all.csv ├── 20230428_3Cboth34_1_C2_int.csv ├── 20230428_3Cboth34_1_C3_all.csv └── 20230428_3Cboth34_1_C3_int.csv
それぞれのサイクルで必要なBB情報がcsvに書き出されています。intはインターナルの略です(今回の場合はallと同じもの)。含まれる情報はシンプルで、以下のようになっています。
% head -n 5 design1/20230428_3Cboth34_1_C3_all.csv smiles,id CC(C)CNCC(C)O,9:1_Test:test_420 CC(C)CNCCCO,9:1_Test:test_287 CN[C@H]1CCCC[C@@H]1O,9:1_Test:test_88 OCCNCc1cccs1,10:1_Test:test_320
sample_generator.py
こちらも機能としては有用なスクリプトなのですが、如何せん使い勝手が悪いです。。上記e_bb_retriever.pyで取り出したサイクル毎のBB情報を使って、全体構造を発生させることができるようです。しかし、各サイクルで使う連結反応や脱保護反応をSMARTS形式で指定する必要があるようで、省略もできません。eDESIGNERで使っているrxnはSMARTSとは異なる記法で書かれており、変換する術を見つけられていません。よって、挙動を試すことも出来ていません。。
bbt_analyzer.py
BBTオブジェクトを解析してくれるスクリプトのようです。/github/edesigner_core/edesignerにいるとして、以下のように使います。
% python bbt_analyzer.py -ts 20230428 bbt_analyzer.v.9.0.0 reading BBTs... performing nbbs by natoms and multi analysis... performing nbbs by natoms and multi analysis... Running time: 0.1 sec. OK % tree -F data data/ ├── 20230428_3Cboth34_0.pic ├── 20230428_3Cboth34_1.pic ├── 20230428_3Cboth34_2.pic ├── 20230428_3Cboth34_3.pic ├── 20230428_3Cboth34_lib_id_list.pic ├── 20230428_BBT_report.csv ├── 20230428_BBTs.pic ├── 20230428_nbbs_by_bbt_and_multi_analysis.csv └── 20230428_nbbs_by_natoms_and_multi_analysis.csv
dataフォルダの下に二つのファイル(_nbbs_)が出力されています。
% head -n 5 ./data/20230428_nbbs_by_bbt_and_multi_analysis.csv bbt index,bbt name,source,multiplicity,number bbs,log(number bbs),order 16,NO_FG;NO_FG;SEC_BOC,ALL,1,1,0.0,0.0 19,NO_FG;NO_FG;calc_ARYLIODIDE,ALL,1,1,0.0,1.0 22,NO_FG;NO_FG;calc_AMINES_AROMATIC_PRIMARY,ALL,1,2,0.3010299956639812,2.0 14,NO_FG;NO_FG;ESTERS_METHYL_ETHYL,ALL,1,3,0.47712125471966244,3.0 % head -n 5 ./data/20230428_nbbs_by_natoms_and_multi_analysis.csv number atoms,multiplicity,source,number bbs 8,1,ALL,0 9,1,ALL,3 10,1,ALL,5 11,1,ALL,13
lib_bb_analyzer.py
BBデータの分析結果を出力するスクリプトです。
% python lib_bb_analyzer.py -ts 20230428 -tg 3Cboth34 lib_bb_analyzer.v.9.0.0 Reading bbts.. Reading lib designs... analyzing lib designs... analyzing bb usage... lib_bb_analyzer.py:169: RuntimeWarning: invalid value encountered in divide times_array = bbs_array / ubbs_array 96 Running time: 0.1 sec. OK % tree -F ./data ./data/ ├── 20230428_3Cboth34_0.pic ├── 20230428_3Cboth34_1.pic ├── 20230428_3Cboth34_2.pic ├── 20230428_3Cboth34_3.pic ├── 20230428_3Cboth34_bb_analysis.csv ├── 20230428_3Cboth34_deprotection_analysis.csv ├── 20230428_3Cboth34_lib_id_list.pic ├── 20230428_3Cboth34_reaction_analysis.csv ├── 20230428_BBT_report.csv ├── 20230428_BBTs.pic ├── 20230428_nbbs_by_bbt_and_multi_analysis.csv └── 20230428_nbbs_by_natoms_and_multi_analysis.csv
dataフォルダにanalysisと付くcsvファイルが三つ作られています。
% head -n 5 ./data/20230428_3Cboth34_bb_analysis.csv number atoms,multiplicity,source,number of BBs,number of unique BBs,unique BBs usage 8,all,ALL,36,1,36.0 9,all,ALL,597,16,37.3125 10,all,ALL,547,20,27.35 11,all,ALL,624,28,22.285714285714285 % head -n 5 ./data/20230428_3Cboth34_deprotection_analysis.csv rindex,count,reaction name 3,54,6.1.1_N-Boc_deprotection_FROM_boc_AND_Null.rxn 6,30,9.7.61_Ester_hydrolysis_FROM_esters_methyl_ethyl_AND_Null.rxn % head -n 5 ./data/20230428_3Cboth34_reaction_analysis.csv rindex,count,reaction name 27,60,2.1.2_Carboxylic_acid_+_amine_condensation_FROM_carboxylic_acids_AND_amines.rxn 26,54,2.1.2_Carboxylic_acid_+_amine_condensation_FROM_amines_AND_carboxylic_acids.rxn 61,30,9.7.8_Amino_to_guanidino_FROM_amines_aliphatic_AND_amines_aliphatic.rxn 3,24,1.3.1_Bromo_Buchwald-Hartwig_amination_FROM_arylbromide_AND_amines_aromatic.rxn
incompatibility_mapper.py
官能基、反応の組み合わせが共存可能かのリストを出力するスクリプトです。
% python incompatibility_mapper.py incompatibility_mapper.v.1.0.0 type "incompatibility_mapper.py -h" for help Importing modules... incompatibility_mapper.v.1.0.0 creating FG incompatibility fileincompatibility... creating reaction incompatibility fileincompatibility... creating deprotection incompatibility fileincompatibility... Running time: 0.0 min. OK % tree -F ./data ./data/ ~~~ ├── deprotection_incompatibility.txt ├── fg_incompatibility.txt └── reaction_incompatibility.txt
dataフォルダ下に_incompatibility.txtというファイルが3つ作成されます。
% head -n 5 ./data/deprotection_incompatibility.txt Deprotection FG onDNAincompatible 0.0.0_NO_REACTION_FROM_Null_AND_Null NO_FG 0 0.0.0_NO_REACTION_FROM_Null_AND_Null ALDEHYDES 0 0.0.0_NO_REACTION_FROM_Null_AND_Null ALKYNES_TERMINAL 0 0.0.0_NO_REACTION_FROM_Null_AND_Null AMINES_ALIPHATIC_SECONDARY 0 % head -n 5 ./data/fg_incompatibility.txt FG1 FG2 incompatible NO_FG NO_FG 0 NO_FG ALDEHYDES 0 NO_FG ALKYNES_TERMINAL 0 NO_FG AMINES_ALIPHATIC_SECONDARY 0 % head -n 5 ./data/reaction_incompatibility.txt Reaction FG onDNAincompatible offDNAincompatible 0.0.0_NO_REACTION_FROM_Null_AND_Null NO_FG 0 0 0.0.0_NO_REACTION_FROM_Null_AND_Null ALDEHYDES 0 0 0.0.0_NO_REACTION_FROM_Null_AND_Null ALKYNES_TERMINAL 0 0 0.0.0_NO_REACTION_FROM_Null_AND_Null AMINES_ALIPHATIC_SECONDARY 0 0
ほぼ備忘録のような形になりましたが、これで一通りeDESIGNERの機能を網羅しました。有効活用していきたいですね。
eDESIGNERを使ってみる(実行編)

前回までに引き続き、オープンソースのDELツールであるeDESIGNERの使い方です。今回はいよいよ本体のスクリプトを実行していきます。環境構築とBBT_input.txtファイルの作成はしてあるものとします。やり方は前2回の記事でまとめています。
DNA Encoded Library (DEL)用のOSSであるeDESIGNERの使い方 - tky_cowのブログ
eDESIGNERを使ってみる(前処理編) - tky_cowのブログ
作業ディレクトリの構成
eDESIGNERのgithubレポジトリを手元にクローンし、/edesigner_core/edesigner/ で作業していくものとします。フォルダ構成は以下のようになっています。
%tree -F -d . ├── BBprep ├── classes │ └── __pycache__ └── resources
BBprepというフォルダを作成し、その中でBBT_input.txtファイルを作成してあります。
BBprep/ ├── BBT_input.txt ├── prepare_bbt_input.py ├── run.sh* ├── sample_annot.txt └── sample_bb.smi
resourcesフォルダには設定ファイルが保存されており、まずはこちらの編集を行います。
resources/ ├── EDESIGNER_PAR_publication_version.v.10.xlsx* ├── antifg.par* ├── calcfg.par* ├── db.par* ├── deprotection.par* ├── enum_deprotection.par* ├── enum_reaction.par* ├── fg.par* ├── headpieces.par* ├── par.par* ├── path.par* └── reaction.par*
設定ファイルの編集
resources以下に入っている3つのファイル(db.par, par.par, path.par)を以下のように編集します。設定ファイルがエディターなどでもかなり見難いので、jsonやxmlにして欲しい所です。。
par.par
このファイルでは矢印で示した5箇所を編集します。何サイクルのDELかで数値を調整する必要があります。下から二番目ではBBTの出力先を指定し、一番下の反応定義はLillyMol内にあるのでそちらを指定します。

path.par
こちらでは途中の作業フォルダ名を設定します。実行した日付と、中間値に設定した重原子数を付けていたようなのでそれに従っています。自動で日付がつく部分があるので、日付は実行日にしておいた方が良いようです。

BBTの作成
設定ができたのでいよいよ実行していきます。
conda activate edesigner
# 出力先フォルダ準備
mkdir bbt
# e_bbt_creater.pyの実行
./run_e_bbt_creater.sh
e_bbt_creator.v.9.0.0
Creating db run folder...
Generating BB types...
Reading compound sets...
Reading Test collection ...
Processing chunk 1 with 1000 records...
731 records remained after filtering by antidel FGs
687 records remained after filtering by belonging to a BBT
687 records remained after desalting smiles
687 records remained after removing molecules with incalculable n atoms
687 records remained after removing molecules with incalculable excess n atoms
678 records remained after removing compounds with excess natoms
678 records remained after pooling chunks
A total of 678 records before combination and elimination of duplicates
A total of 678 records remained after combination and elimination of duplicates
Updating BBTs...
Reporting compound files...
pickling BBTs object...
Reporting BBTs file...
Run time = 0.0 min
OKpandas関係のwarningが出たりしますが、上記のような表示が出ていれば成功です。compsとdataフォルダが作られ、以下のようなファイルができていると思います。
% tree -F comps
comps/
└── 20230428/
├── 1.smi
├── 124.smi
├── 128.smi
├── 130.smi
├── 132.smi
├── 133.smi
├── 136.smi
~~~~~~~~略~~~~~~~~~
% tree -F data
data/
├── 20230428_BBT_report.csv
└── 20230428_BBTs.pic
eDESIGNsの作成
続いて論文中で言う所のeDESIGNsを作成するためには、e_designer.pyを実行します。run_とついている方を使ってもいいですが、run_*系のスクリプトは所定のフォルダが作られているか確認した後に、pythonスクリプトを実行するだけですので、python直接実行でも変わりません。
% python e_designer.py
e_designer.v.9.0.0
Loading BBTs...
Creating reaction indexes...
Creating designs...
Creating headpieces...
this round number designs = 6
cycle 0 number designs = 6
Saving designs to file...
Creating cycle 1...
Reading from disk from design number 0...
Processing 6 designs...
Time to expand designs: 1.0 seconds
Writting to disk 271 designs...
cycle 1 number designs = 271
Creating cycle 2...
Reading from disk from design number 0...
Processing 271 designs...
Time to expand designs: 1.0 seconds
Writting to disk 5622 designs...
cycle 2 number designs = 5622
Creating cycle 3...
Reading from disk from design number 0...
Processing 5622 designs...
Time to expand designs: 4.0 seconds
Removing desings with not enough size...
Adding lib_id to designs...
Maintaining lib_id dictionary...
Adding desing_id to desings...
Writting to disk 38736 designs...
cycle 3 number designs = 46659
Numebr of designs after removing low density desings: 38736
Number of library ids generated: 3065
Saving lib_id_list to file...
Running time: 0.1 min.
OKこんな表示になれば成功です。この例ではBBが1000個入ったデータを使いましたが、可能な3サイクルのDELは結構な数になることが見て取れます。このツールを使う利点の一つでもあると思います。dataフォルダにファイルが増えていることを確認できます。
% tree -F data data/ ├── 20230428_3Cboth34_0.pic ├── 20230428_3Cboth34_1.pic ├── 20230428_3Cboth34_2.pic ├── 20230428_3Cboth34_3.pic ├── 20230428_3Cboth34_lib_id_list.pic ├── 20230428_BBT_report.csv └── 20230428_BBTs.pic
libDESIGNsの作成
次に、lib_designer.pyを使ってlibDESIGNsを作っていきます。ここも実行するだけです。
% python lib_designer.py lib_designer.v.9.0.0 Loading BBTs... Loading dictionary of libraries... Creating libraries for 3065 library ids Reading designs... Processing libraries... Processing 3065 libraries... 72 libraries remaining after removing those that do not fulfill the criteria Saving libraries to disk... Running time: 0.6 min. OK % tree -F results results/ └── 20230428_3Cboth34_libDESIGNS.pic
ここで候補のライブラリー情報が読み取られ、par.parに記載した条件を満たすようなライブラリーのみに絞り込まれます。結果は、上に示したように、resultsフォルダ下にpickleオブジェクトとして保存されます。
library configの出力
最終ステップとして、lib_design_interpreter.py を使って各ライブラリーのレシピ情報のようなファイルを作成します。
% python lib_design_interpreter.py lib_design_interpreter.v.9.0.0 Loading BBTs... Loading pickled designs to object... Creating config.txt file... Translated 72 library designs Running time: 0.0 min. OK % tree -F results results/ ├── 20230428_3Cboth34_config.txt └── 20230428_3Cboth34_libDESIGNS.pic
フィルターを通過した72のライブラリー情報が変換され、resultsフォルダに_configとして書き出されます。中身は以下のようになっており、全てのライブラリー情報が一つのファイルにまとまっていますので、特定の情報を抜き出すにはパースする必要があります。

Start enumeration instructionsからEnd enumeration instructionsまでが一つのDELの情報となっており、今回の場合は72種のDEL情報が一つのファイルに含まれています(.INTERNALと.ALLは同じなので144)。INTERNALのBBとEXTERNALを分けて準備した場合別に使えるようにという配慮のようです。
どのように活用するか
ここまででeDESIGNERの基本的な機能を使いましたので、どのように活用できそうか考えてみます。
- トータルバランスの悪いBBの除外
- 新たに作るライブラリー案のアイデア出し
- 作る前に構造発生して物性値分布の確認
トータルバランスの悪いBBの除外
設定した条件を満たすようなBB、ライブラリーが残ってくるのでフィルターとして使えます。
新たに作るライブラリー案のアイデア出し
反応やBBの組み合わせが網羅的に出てきますので、内容を読み取れば意外なBBや反応の組み合わせが見つかることがあります。
作る前に構造発生して物性値分布の確認
ここまでの機能だけではできませんが、configに従って構造発生することで事前に物性値を計算することができます。
まとめ
記事3つ分かかってしまいましたが、eDESIGNERの使い方を見てきました。本ツールに限らず、オープンソースツールを使って色々なモダリティに手を出すことができるようになってきています。うまく活用できるようになっていきたいなと思います。
eDESIGNERを使ってみる(前処理編)

前回の記事から大分間が空いてしまいましたが、引き続きオープンソースツールのeDESIGNERの使い方を書いていきます。今回は、eDESIGNERで使う試薬(Building Block, BB)ファイルはどのような形で用意すれば良いかという所までを扱います。eDESIGNERの概要と環境構築については前記事をご覧ください。
tky-pychemweb.hatenablog.com
なお、前回の環境構築はLinuxで行いましたが、eDESIGNER自体はpythonのみで動作し、M1チップのMBAでも動作確認が取れたため、スクショはMacで撮っています。
eDESIGNER全体の流れ
eDESIGNERが行う処理の全体の流れを下図に示します。

ライブラリーを構成するBBのリストからスタートし、含まれる化合物の全体構造を得ることをゴールとすると、eDESIGNERが担当するのは、アノテーション付きのBBファイルを入力にして、ライブラリーを構築するレシピのような設定ファイルを出力するところまでになります。その前後に当たるアノテーション付きのBBファイルの準備と構造発生で、LillyMolが必要になるところが使うのを難しくしている要因です。そしてお試しのサンプルファイルもないんですよね。。この「アノテーション付きのBBファイル」というのは、どのようなファイルをどうやって準備すれば良いのか、というのが今回のメインテーマです。
アノテーション付きのBBファイル
アノテーションとは何ぞやという点ですが、各BBがどのような官能基をいくつ持っているかを数値の羅列で表現したファイルになります。何の官能基を持っているかを調べてくださいというのが事前に定められており、それに従ってファイルを準備する必要があります。さらに、eDESIGNER処理で利用するID、分子量、回転可能結合数も追記しています。このファイルを準備することが、今回のゴールになります。
アノテーション付きファイルの作成
入力BBファイル
まず、入力となるBBのファイルをSMILES形式で準備します。この段階では分類などをする必要はなく、ただ使うかもしれないBBを全て含むファイルを作れば大丈夫です。RDKitで読み込めないと今後エラーが出るので、この段階でRDKitで準備しておくとスムーズでしょう。このファイルをsample_bb.smiというファイルとします。
tsubstructure
元論文のSupplementary informationをよく読むとLillyMolのtsubstructureというツールを使うと書かれています。>
このまま使うとうまくいかない部分があるため、オプションを追加して以下コマンドを実行します。username部分は各自のインストールフォルダに合わせて変更してください。
tsubstructure -v -u -q F:/home/<username>/github/LillyMol/contrib/data/DEL/AnnotationQueries/queries_del -A D -a ./sample_bb.smi > ./sample_annot.txt
この結果出力されるsample_annot.txtは以下のようなファイルになります。40-50の定義された官能基を各BBがいくつ有しているかを羅列したファイルが出来上がります。
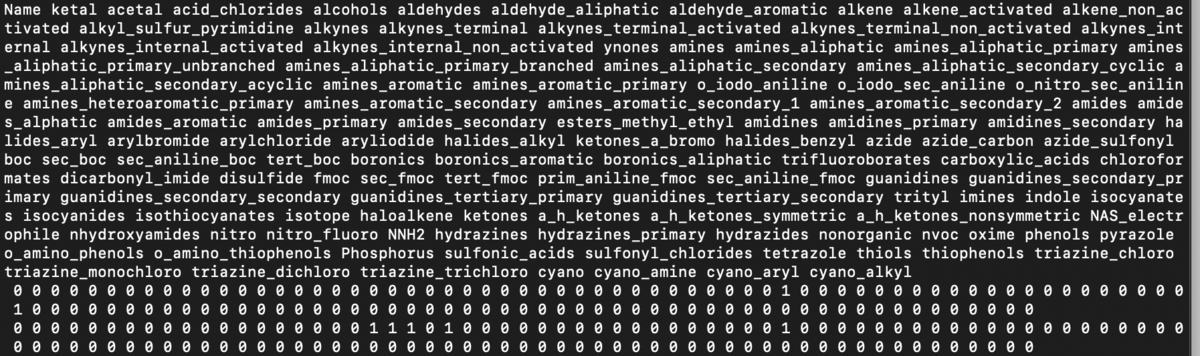
統合ファイルの作成
最後に、IDといくつかの物性値を追加して、eDESIGNERのインプットに使うファイルを作成します。sample_bb.smiとsample_annot.txtを統合するpythonスクリプト(prepare_bbt_input.py)を作成して処理します。
python prepare_bbt_input.py sample_bb.smi sample_annot.txt
スクリプトの中身は以下のようになっています。
import sys import pandas as pd from rdkit import Chem from rdkit.Chem import AllChem, Descriptors def calc_props(smiles_list): id_list = [] mw_list = [] rotb_list = [] for idx, smi in enumerate(smiles_list): bbid = f"test_{idx}" mol = Chem.MolFromSmiles(smi) mw = Descriptors.MolWt(mol) rotb = Descriptors.NumRotatableBonds(mol) id_list.append(bbid) rotb_list.append(rotb) mw_list.append(mw) prop_df = pd.DataFrame({"ID": id_list, "SMILES": smiles_list, "ROTB": rotb_list, "MW": mw_list}) return prop_df def merge_annot(prop_df, annot_file): df_annot = pd.read_csv(annot_file, sep=" ") df_annot = df_annot.drop(columns=["Name"]) if df_annot.shape[0] == prop_df.shape[0]: df = pd.concat([prop_df, df_annot], axis=1) else: print("Number of rows is not the same.") exit() df.to_csv("BBT_input.txt", index=False, sep=" ") if __name__ == '__main__': args = sys.argv if len(args) == 3: smiles_file, annot_file = args[1], args[2] smiles_list = [] with open(smiles_file, 'r') as inp: for smi in inp.readlines(): smiles_list.append(smi.rstrip()) prop_df = calc_props(smiles_list) merge_annot(prop_df, annot_file) else: print("Usage: python prepare_bbt_input.py smiles_filename annotation_filename") exit()
calc_propsでIDと物性値を生成し、 merge_annotでアノテーション情報を統合してBBT_input.txtというファイルに出力します。
完成した入力ファイル
以上を実行した結果得られるBBT_input.txtは以下のようなファイルになります。なお、ID部分のカラム名はIDにしておかないと後々エラーが出るようです。次の記事では、いよいよこのファイルを使ってeDESIGNERを動かしていきます。

eDESIGNERを使ってみる(準備編)

最近は製薬業界でもオープンソース、オープンサイエンスの流れが来ており、企業レポジトリからも様々なツールが公開されています。私も仕事柄そのようなオープンなツールを利用したり改変したりして、自分の仕事でも使えないか試行錯誤しています。本エントリーでは、その中の一つであるeDESIGNERを紹介します。軽い気持ちで始めたはいいものの、クソゲーと化した途中非常に苦労したため、使い方を書き残しておこうと思いました。
eDESIGNERとは
eDESIGNERはEli Lilly社の研究員によって開発・公開されたDNA Encoded Library (DEL)用のツールです。ざっくり言うと、重原子数を制御することで物性値をコントロールし、いわゆるドラッグライクなDELを作り出すためのツールです(たぶん)。プログラムだけでなく、論文もオープンアクセスになっているので、興味のある方は元文献を参照ください。
レポジトリ:https://github.com/jamflcgh/edesigner_core
元文献:Navigating the DNA encoded libraries chemical space | Communications Chemistry
DELについて
DELはDNAタグで化合物の識別を可能にした、低分子化合物のライブラリーです。ライブラリーそのものだけでなく、ライブラリーの構築やそれを使ったスクリーニングまでを含めてDELという呼称を使っている場合もあるかと思います。コンセプトとしては、ケミカルバイオロジー版コンビケムライブラリーのようなものとも考えられます。使える反応は限られますが、一つのチューブに数百万~数億化合物のライブラリーを持つことが可能であり、(HTSに比べて)アッセイや管理が簡便になるのが特徴です。海外では5-10年ほど前からスクリーニングの1手法として用いられてきており、最近論文発表が散見されています。
eDESIGNERのアプローチ
DELの難点の一つに、全体的にサイズが大きくなりがちという点があります。よく使われる3cycleのDELでは、rule-of-fiveを大きく逸脱することも少なくありません。また、ライブラリーサイズが数千万ー数億件になることが一般的で、この数になると全化合物の分子量やLogPを計算することも大変になります。そこで彼らは、全体構造を構築せずとも計算可能な重原子数でコントロールすることを試みています。論文データ及び自分でいくつか試した感触ですと、結構うまくワークするようです。
環境構築
前置きが長くなりましたが、環境構築を行っていきましょう。eDESIGNER自体はpythonで書かれたプログラムですが、残念ながら単独で動かすことはできません。インプットとなるビルディングブロック試薬のファイル、ならびに最終的な化合物の構造生成にLillyMolという別プログラムが必要になるため、こちらの準備も行います。
eDESIGNERの環境構築
こちらは難なく構築可能です。UNIX系でminicondaが利用可能である状態であるとします。一応、READMEに書かれているバージョンに揃えるようにしていますが、あまり気にしなくて大丈夫なようです。
cd /home/hogehoge/github/ git clone https://github.com/jamflcgh/edesigner_core.git . conda create env -n edesigner python=3.6.5 conda activate edesigner conda install rdkit=2017.09.3 #上記でpandas, numpyも入ると思います
LillyMolのセットアップ
eDESIGNERと異なりLillyMolはC++ベースで書かれており、ビルドやコンパイルなどと言われるような操作が必要です。成功すると実行可能なバイナリーファイルが100個近く生成されます。私はC++を読むことも書くこともできないせいか、READMEを読んでもインストール方法がさっぱりでしたので、iwatobipen先生の記事を参考に設定しましょう。
レポジトリ:https://github.com/EliLillyCo/LillyMol
# conda環境に入った状態から続ける conda install -c conda-forge zlib eigen3 git clone https://github.com/EliLillyCo/LillyMol.git cd LillyMol # makefile.public.xxxをコピーして書き換える ./makeall.sh
OSとgccのバージョンに合わせてmakefile.public.xxxのファイルをコピーで作成し、ブログに従って内容を書き換えます。makeall.shスクリプトが成功するとLillyMol/bin/Linux-gcc-9.4.0/のようなフォルダが作成され、その下にプログラムが配置されるので、パスを通しておきます。
export PATH=/home/hogehoge/github/LillyMol/bin/Linux-gcc-9.4.0/:$PATH
次は(いつになるか分かりませんが)、実際に使ってみる編に続きます。
